四川大学考古科学中心在法医学Top期刊FSIG报道全球人群non-CODIS STR图谱为法医复杂DNA分析提供新思路

短串联重复序列(Short tandem repeat,STR)约占基因组的6%,是一类高度多态性的遗传标记。自上世纪90年代首次应用在法医DNA分析领域以来,STR在群体遗传学研究、个人识别、生物地理祖先推断、亲子鉴定和亲缘关系分析等领域发挥了重要作用。目前主流的STR检测试剂盒一般仅包含FBI联合DNA索引系统(Combined DNA Index System,CODIS)核心基因座在内的20-30个STR标记,难以满足日益复杂的法医DNA分析需求。高通量新一代测序技术(Next-generation sequencing,NGS)使得单次试验能够联合检测大量CODIS和non-CODIS STR遗传标记,从而有效提高DNA分析体系的系统效能,有望为复杂DNA分析带来新的突破。然而,由于缺乏全球人群non-CODIS STR的遗传变异图景,这一技术路线并未在实际案件中得到广泛应用。
近日,四川大学华西医院罕见病研究院和四川大学考古科学中心何光林课题组联合广东省毒品实验技术中心中国工程院刘超院士团队在法医学权威期刊Forensic Science International: Genetics在线发表了题为《Comprehensive landscape of non-CODIS STRs in global populations provides new insights into challenging DNA profiles》的研究论文,利用来自全球不同群体的4150个高深度全基因组测序(Whole genome sequencing,WGS)数据对178个non-CODIS STR的多态性特征进行了全面解析。在此基础上,该研究结合真实和模拟数据集,对大规模non-CODIS STR体系解决多贡献者的混合DNA分析、混合遗传背景下的生物地理祖先推断和复杂亲缘关系鉴定等法医学难点问题的应用潜力进行了系统性评估。该研究首次揭示了全球群体基因组non-COIDS STR的多态性模式,证明纳入多态性的non-CODIS STR对于提升复杂DNA分析能力具有重要意义,为后续大规模STR检测体系的开发提供了科学依据。
研究成果:
1.全基因组non-CODIS STR参考基因集构建
图1.研究纳入的STR基因座位染色体分布。
研究共纳入198个多态性STR位点,包括20个CODIS核心基因座和178个non-CODIS基因座,并通过序列搜索、重复序列比对和手动核对,完善了全部STR的基因组注释信息。
2.全球群体non-CODIS STR多态性和法医学应用潜力评估
图2. CODIS和non-CODIS多态性和法医学参数评估。a:全球群体STR多态性;b:STR法医学应用参数。PIC,多态性信息量(Polymorphism Information Content);He,期望杂合度(Expected heterozygosity);Ho,观察杂合度(Observed heterozygosity);MP,匹配概率(Match probability);PD,个人识别概率(Power of discrimination);PE,排除概率(Power of exclusion);TPI,典型父权指数(Typical paternity index)。
该研究对STR在不同洲际群体的多态性和法医学参数进行了评估。结果表明non-CODIS STR具有与CODIS STR相当的多态性和法医学效能。此外, CODIS和non-CODIS STR均表现出明显的群体分层:非洲群体(AFR)总体多态性较高,而大洋洲群体(OCN)多态性最低。
3. 群体结构和生物地理祖先推断
在考虑群体多态性和连锁不平衡模式的情况下,该研究模拟了包含20个CODIS核心基因座和88个non-CODIS基因座的大规模STR检测体系(extdSTR),实现了对全球不同洲际群体遗传背景的有效区分。
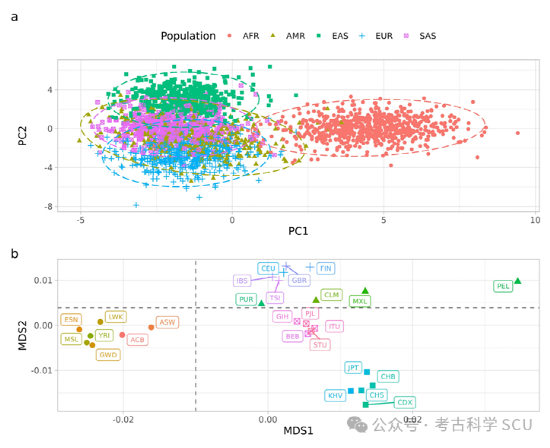
图3. 基于108个STR的全球群体遗传结构分析。a:五大洲际群体PCA分析;b:全球26个群体基于群体分化指数(Fst)矩阵的非度量多维尺度分析。

图4. 全球26个群体STRUCTURE非监督聚类分析(K=4,5,6)。
进一步,该研究构建了基于108个STR的随机森林分类模型,用以评估体系的生物地理祖先推断能力。结果表明,应用大规模non-CODIS STR可以有效区分不同洲际祖源背景个体的生物地理祖先。
表1 基于extdSTR和CODIS核心位点集预测祖先来源的RF模型混淆矩阵。TP:真阳性;FP:假阳性。

4. 混合DNA解析
为验证non-CODIS对于复杂混合DNA的解析能力,该研究模拟生成了一系列由2到8个贡献者构成的混合DNA图谱。研究结果表明,纳入non-CODIS STR有效提升了贡献者人数(NoC)估计的准确性和混合DNA图谱的证据强度。

图5. 使用extdSTR和CODIS核心位点集对DNA混合图谱进行反卷积(n = 100)。真实贡献者人数NoC为2-8人。a:NoC推断准确性评估;b:混合DNA图谱中目标个体的似然率值。
5. 复杂亲缘关系推断
最后,该研究评估了non-CODIS进行复杂亲缘关系推断的能力。结果表明,大规模non-CODIS能够实现对一到三级亲缘关系的有效区分,但对于三级以上亲缘关系区分能力仍然欠佳。

该研究利用高深度WGS数据对全球群体基因组non-CODIS STR的多态性特征和法医学应用潜力进行了系统性分析。研究结果证明non-CODIS STR在不同群体均具有较高的多态性和法医学效能,在生物地理祖先推断、DNA混合反卷积和复杂亲缘关系分析等领域具有广阔的应用前景。随着NGS技术在法医学领域的广泛应用,将non-CODIS STR纳入大规模STR检测体系有望为复杂DNA分析提供强有利的工具。
论文第一作者为四川大学华西医院罕见病研究院博士后黄雨果,第一通讯作者为四川大学华西医院罕见病研究院和四川大学考古科学中心何光林副研究员,广东省毒品实验技术中心(国家毒品实验室广东分中心)刘超院士为共同通讯作者。


